
近日,国家超级计算长沙中心副主任彭绍亮团队在国际顶级期刊Nature Machine Intelligence发表了题为Multi-task Joint Strategies of Self-supervised Representation Learning on Biomedical Networks for Drug Discovery的研究论文。该研究提出了面向药物发现的生物网络自监督表征学习的多任务联合框架MSSL2drug,系统地研究了如何有效地组合多个自监督模型这一人工智能领域的挑战性问题,探索发现了基于生物网络的多模态网络表征学习技术,为缺乏生物或临床标注数据的药物发现提供了新的研究思路。

传统的药物研发周期长、费用高,成功低。近些年来,面向生物医学网络的图深度学习技术在药物发现领域中显示出了广阔的前景。然而,生物医学数据的稀疏性和高度复杂性限制了图深度学习技术在特定药物发现中的性能。该研究组经过调研发现国内外研究者对多任务驱动的自监督表征学习技术缺少全面系统的了解,尤其在药物发现领域中往往局限于单任务驱动的自监督表征技术,如何有效地组合多个自监督模型充满着挑战。为了缓解这些痛点,提出了MSSL2drug技术体系,主要基于生物医学异构网络的结构、语义和属性等多模态特征,设计了6个单任务和15种多任务组合,开发了基于图注意力的对抗多任务学习框架生成分子表征,并系统地评估不同的多任务自监督模型在药物发现应用中的差异性。最终发现多模态的自监督任务组合能够获得最高的药物预测性能;如果当模态信息相同时,局部-全局任务组合优于随机的任务组合。与基准方法相比,本文发现的多模态表征模型在不同的测试场景中获得更高的精度。
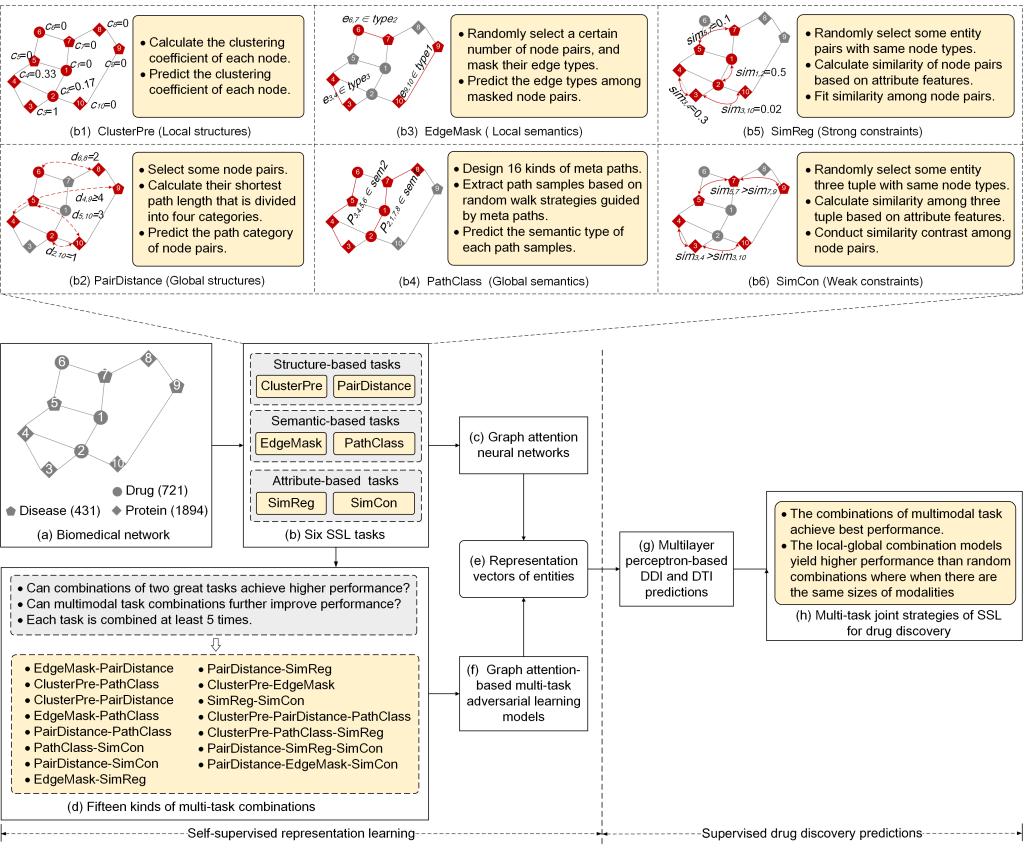
MSSL2drug研究框架
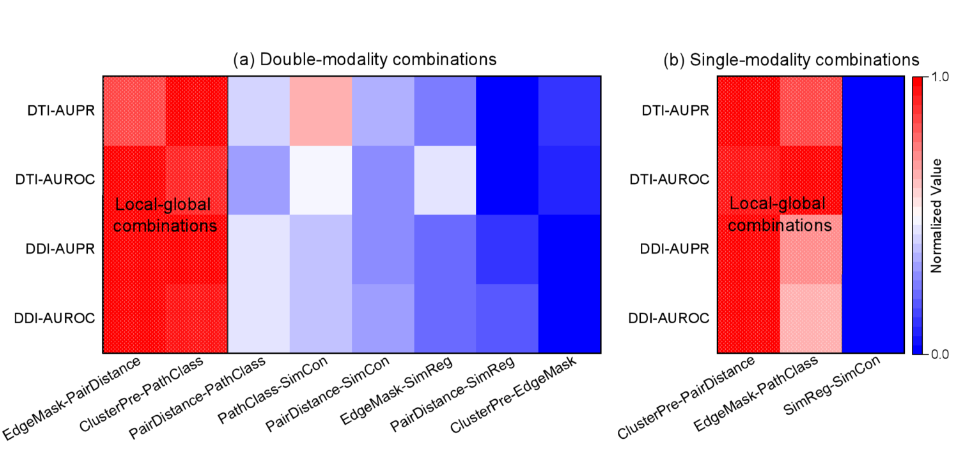
同一模态下,局部-全局自监督任务组合优于随机任务组合
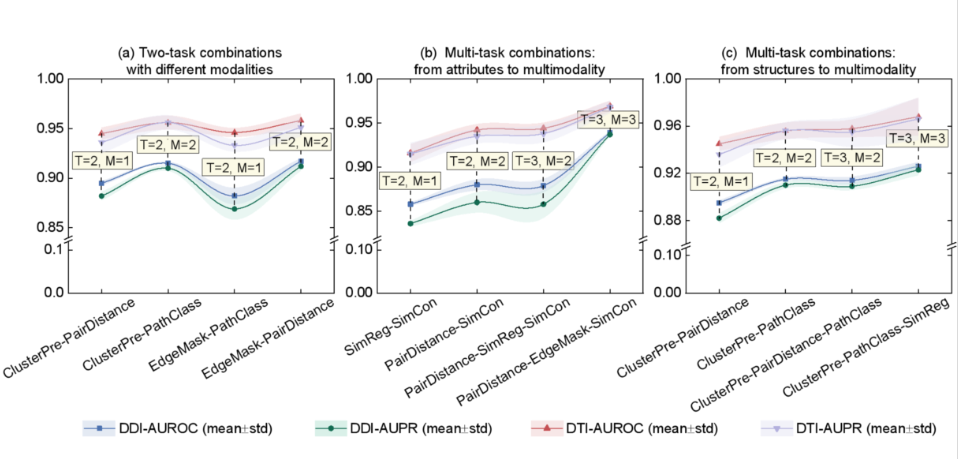
多模态的自监督任务组合可以获得最高的性能
该研究关注抗炎症风暴药物研发的迫切需求,利用MSSL2drug进行抗炎症因子IL-6蛋白的药物筛选,并通过PubMed生物医学文献、临床报告、分子动力学模拟和表面等离子共振(SPR)等多维数据表明MSSL2drug预测的vandetanib (KD=28.6μM)和pazopanib (KD=20.7μM)可以与IL-6蛋白高亲和力结合,对加速抗炎症药物研发具有重要的意义。
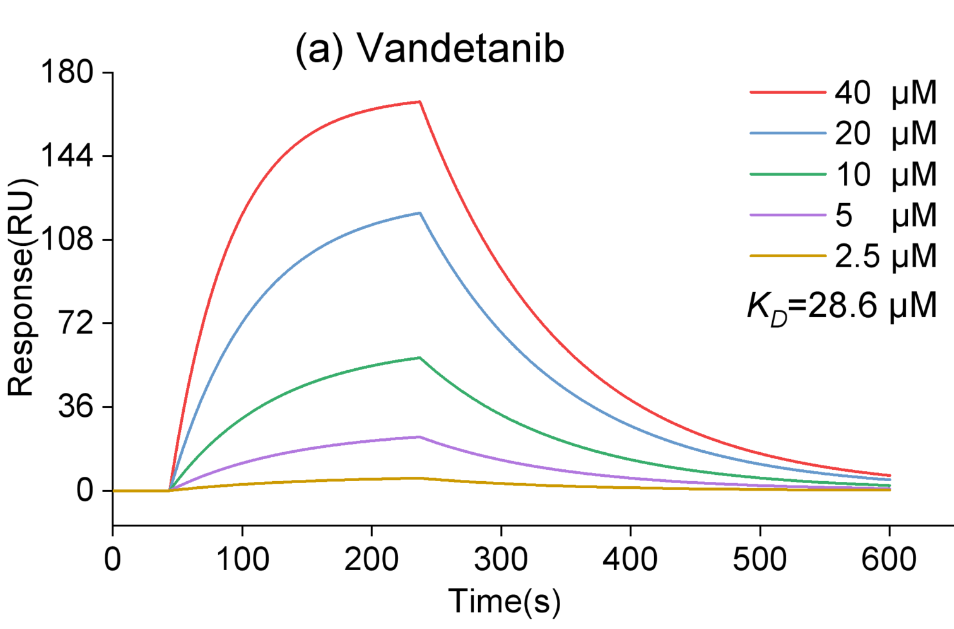
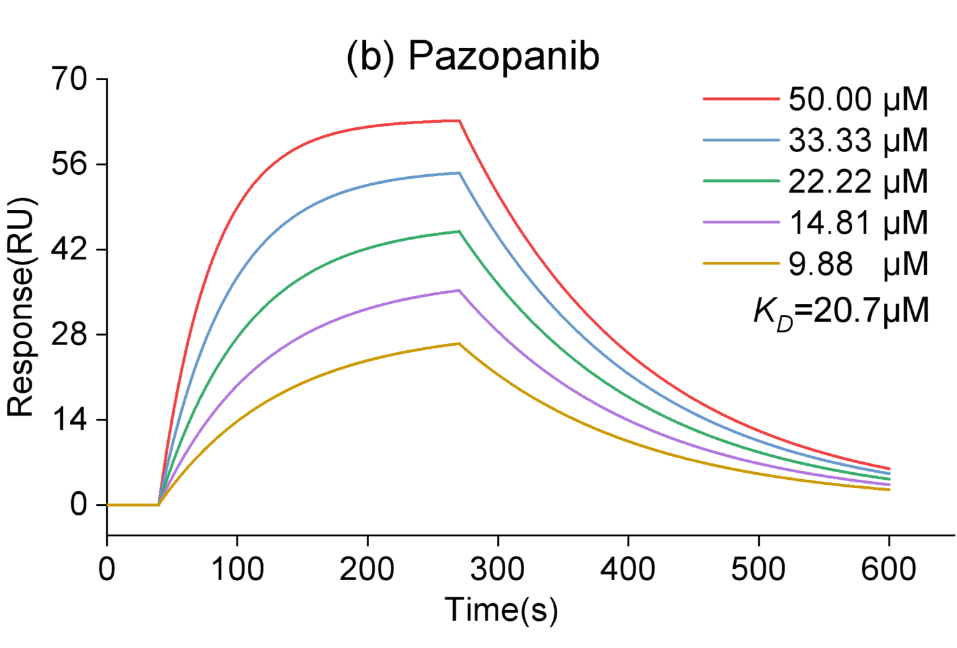
表面等离子共振技术验证候选药物与靶标蛋白的亲和力
该研究论文湖南大学博士研究生王小奇为第一作者,超算中心彭绍亮副主任和中国科学院计算机网络信息中心李非副研究员为论文通讯作者。研究受到国家自然科学基金支持。国家超级计算长沙中心和鹏城实验室为该研究的开展提供了强大的算力支持。
论文地址:https://www.nature.com/articles/s42256-023-00640-6